Os transformadores são uma grande tendência na visão computacional. Recentemente, dei uma visão geral de algumas incríveis Avanços. Desta vez, usarei minha reimplementação de um modelo baseado em transformador para segmentação 3D. Em particular, usarei o famoso transformador UNTR e tentarei ver se ele é parecido com um clássico. O caderno está disponível.
UNRETRA é a primeira arquitetura de transformador bem -sucedida para a segmentação de imagem médica 3D. Nesta postagem do blog, tentarei corresponder aos resultados de um Sonhos Modelo no conjunto de dados BRATS, que contém imagens de cérebro de ressonância magnética 3D. Aqui está uma visão geral de alto nível da UNRET que treinaremos neste tutorial:
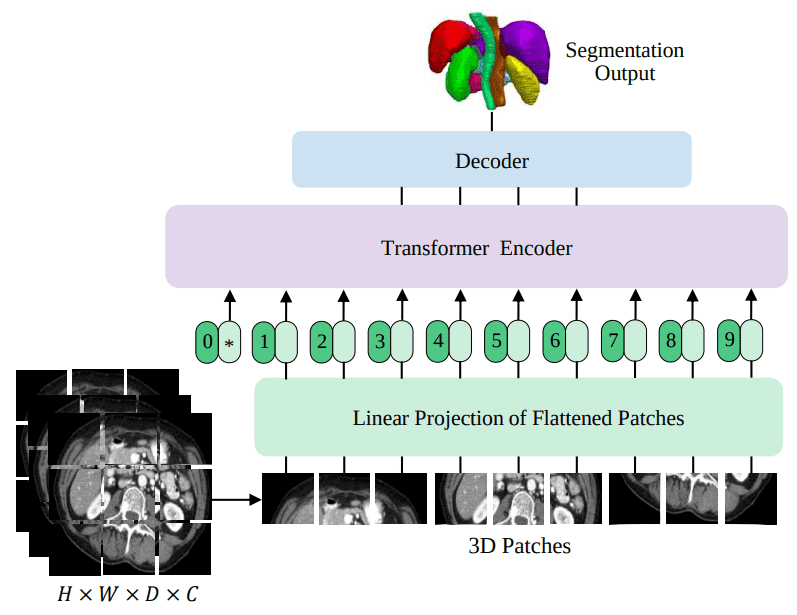
Fonte: UNRET: Transformadores para segmentação de imagem médica 3D, Hatamizadeh et al.
Para testar minha implementação, usei um tutorial existente em um conjunto de dados de segmentação de ressonância magnética 3D. Assim, tenho que dar crédito à incrível biblioteca de código aberto da NVIDIA chamado Mona por fornecer o tutorial inicial que modifiquei para fins educacionais. Se você gosta imagem médica Não deixe de conferir esta biblioteca incrível e seus tutoriais.
Vamos ver os dados primeiro!
ATUALIZAÇÃO: Lançamento do livro! Aprenda sobre “Aprendizado profundo em produção”Para atender seus modelos de ML a milhões de usuários.
Conjunto de dados dos pirralhos
Pirralhos é um conjunto de dados de imagem 3D em larga escala multimodal. Ele contém 4 volumes 3D de imagens de ressonância magnética capturadas sob diferentes modalidades e configurações. Aqui está uma amostra do conjunto de dados. É importante ver que apenas o tumor está anotado. Isso torna as coisas como a segmentação mais difícil, pois o modelo precisa se localizar no tumor.
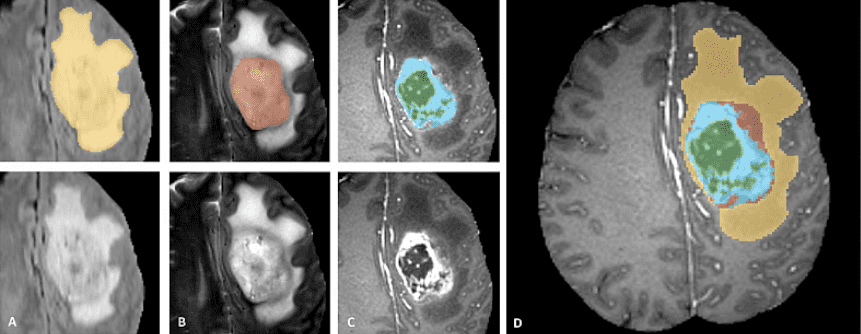
Imagem oficial de teaser de dados do site de conclusão dos pirralhos
Os patches de imagem representam categorias de tumores da seguinte forma (da esquerda para a direita):
-
Edema: Todo o tumor (amarelo) é geralmente visível na imagem de ressonância magnética do Flair T2.
-
Núcleo sólido que não aumenta: O núcleo do tumor (vermelho) visível na ressonância magnética T2.
-
O Melhorando o tumor estruturas (azul claro). Geralmente visível em T1GD, ao redor do núcleo necrótico (verde).
-
As segmentações são combinadas para gerar os rótulos finais do conjunto de dados.
Com MONAIcarregando um conjunto de dados do Concorrência de decatlo de imagem médica torna -se trivial.
Carregamento de dados com monai e transformações
Utilizando o DecathlonDataset Classe de Monai Library, pode carregar qualquer um dos 10 conjuntos de dados disponíveis no site. Vamos usar Task01_BrainTumour no nosso caso.
cache_num = 8
from monai.apps import DecathlonDataset
train_ds = DecathlonDataset(
root_dir=root_dir,
task="Task01_BrainTumour",
transform=train_transform,
section="training",
download=True,
num_workers=4,
cache_num=cache_num,
)
train_loader = DataLoader(train_ds, batch_size=2, shuffle=True, num_workers=2)
val_ds = DecathlonDataset(
root_dir=root_dir,
task="Task01_BrainTumour",
transform=val_transform,
section="validation",
download=False,
num_workers=4,
cache_num=cache_num,
)
val_loader = DataLoader(val_ds, batch_size=2, shuffle=False, num_workers=2)
Importações e funções de suporte podem ser encontradas no caderno. O que é crucial aqui é o oleoduto de transformaçãoque garanto não é uma coisa fácil nas imagens 3D. MONAI Fornece algumas funções para fazer um pipeline rápido para os fins deste tutorial. Detalhes como o Orientação da imagem são deixados de fora do tutorial de propósito.
Resumidamente, resservamos nossas imagens a um tamanho de voxel de 1,5, 1,5 e 2,0 mm em cada dimensão. Posteriormente, tomamos sub-volumes 3D aleatórios de tamanhos 128, 128, 64. Isso, obviamente, precisa ser aplicado à imagem de entrada e ao Segmentação máscara.
Em seguida, são aplicados alguns aumentos, como virar aleatoriamente o primeiro eixo e redimensionar a intensidade (treming).
A classe ConvertToMultiChannelBasedOnBratsClassesd Traz os rótulos para o formato que queremos.
from monai.transforms import (
Activations,
AsChannelFirstd,
AsDiscrete,
CenterSpatialCropd,
Compose,
LoadImaged,
MapTransform,
NormalizeIntensityd,
Orientationd,
RandFlipd,
RandScaleIntensityd,
RandShiftIntensityd,
RandSpatialCropd,
Spacingd,
ToTensord,
)
roi_size=(128, 128, 64)
pixdim=(1.5, 1.5, 2.0)
class ConvertToMultiChannelBasedOnBratsClassesd(MapTransform):
"""
Convert labels to multi channels based on brats classes:
label 1 is the peritumoral edema
label 2 is the GD-enhancing tumor
label 3 is the necrotic and non-enhancing tumor core
The possible classes are TC (Tumor core), WT (Whole tumor)
and ET (Enhancing tumor).
"""
def __call__(self, data):
d = dict(data)
for key in self.keys:
result = ()
result.append(np.logical_or(d(key) == 2, d(key) == 3))
result.append(
np.logical_or(
np.logical_or(d(key) == 2, d(key) == 3), d(key) == 1
)
)
result.append(d(key) == 2)
d(key) = np.stack(result, axis=0).astype(np.float32)
return d
train_transform = Compose(
(
LoadImaged(keys=("image", "label")),
AsChannelFirstd(keys="image"),
ConvertToMultiChannelBasedOnBratsClassesd(keys="label"),
Spacingd(
keys=("image", "label"),
pixdim=pixdim,
mode=("bilinear", "nearest"),
),
Orientationd(keys=("image", "label"), axcodes="RAS"),
RandSpatialCropd(
keys=("image", "label"), roi_size=roi_size, random_size=False),
RandFlipd(keys=("image", "label"), prob=0.5, spatial_axis=0),
NormalizeIntensityd(keys="image", nonzero=True, channel_wise=True),
RandScaleIntensityd(keys="image", factors=0.1, prob=0.5),
RandShiftIntensityd(keys="image", offsets=0.1, prob=0.5),
ToTensord(keys=("image", "label")),
)
)
val_transform = Compose(
(
LoadImaged(keys=("image", "label")),
AsChannelFirstd(keys="image"),
ConvertToMultiChannelBasedOnBratsClassesd(keys="label"),
Spacingd(
keys=("image", "label"),
pixdim=pixdim,
mode=("bilinear", "nearest"),
),
Orientationd(keys=("image", "label"), axcodes="RAS"),
CenterSpatialCropd(keys=("image", "label"), roi_size=roi_size),
NormalizeIntensityd(keys="image", nonzero=True, channel_wise=True),
ToTensord(keys=("image", "label")),
)
)
É sempre melhor ver o oleoduto em ação, visualizando algumas fatias de todas as modalidades. Abaixo está uma amostra de nossos dados de trem:
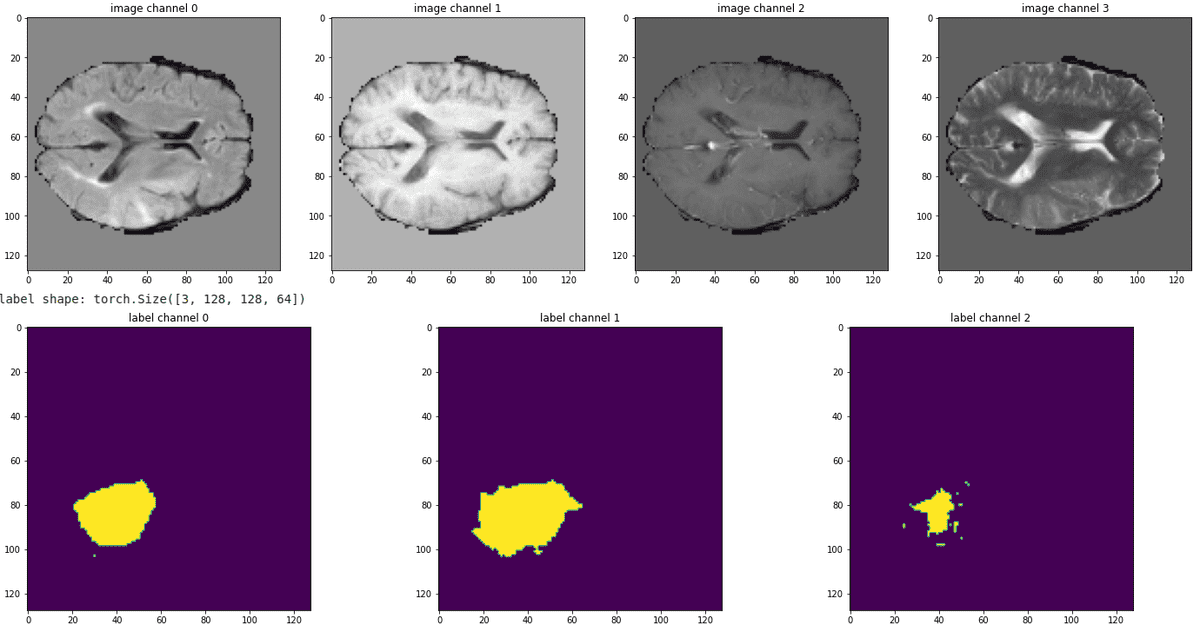
Fonte: Imagem do autor com base no caderno
Pode -se observar que o tumor é não mutuamente exclusivo. Nesse sentido, esperamos que o aprimoramento do tumor e das células necróticas (mapa da segmentação mais à direita) seja o mais difícil de prever.
O pipeline de dados e transformação agora estão todos definidos. Vamos dar uma olhada na arquitetura do modelo.
Saiba mais sobre a IA aplicada em aplicativos de imagem médica no curso bem estruturado Você tem para medicina Oferecido pela Coursera.
A arquitetura UNRETRA
Aqui está a arquitetura do modelo que incorpora transformadores no infame Sonhos arquitetura:
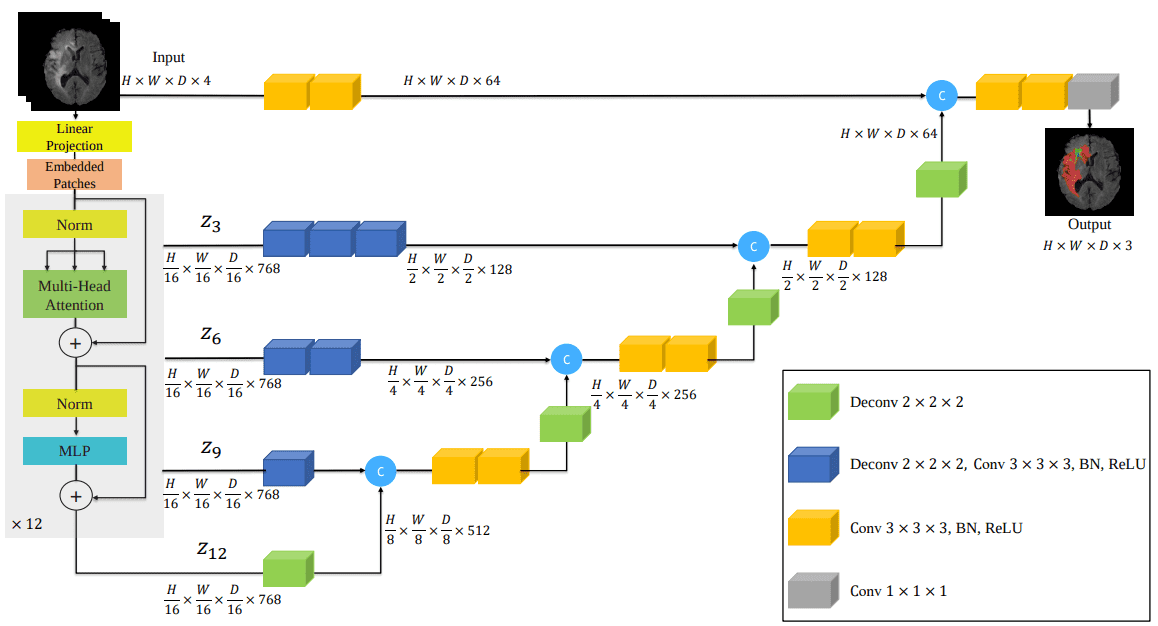
Fonte: UNRET: Transformadores para segmentação de imagem médica 3D, Hatamizadeh et al.
Curiosamente, comecei a implementar esse modelo como na figura do papel representada acima. Mais tarde, descobri que já foi implementado em Monai. Depois de verificar seu código, encontrei detalhes significativos ausentes. Conclusão: Não confie nas imagens da arquitetura, elas não incluem toda a história sobre como implementar o artigo. Para ver o código de implementação, consulte minha implementação no Auto-Attention-CV biblioteca.
Agora posso finalmente usar minha implementação da UNRE. Criei uma pequena biblioteca que implementa vários blocos de auto-distribuição para visão computacional e os embalam em um pacote instalável por PIP. Então agora eu só tenho que instalar meu pacote PIP que contém o modelo e o voila:
$ pip install self-attention-cv==1.2.3
Para inicializar o modelo, precisamos fornecer o tamanho do volume, as modalidades de imagem de entrada, o número de rótulos (output_dim) e várias coisas sobre o Transformador de visão. Os exemplos incluem incorporar dimensão do patch, tamanho do patch, número de cabeças, tipo de normalização etc.
from self_attention_cv import UNETR
device = torch.device("cuda:0")
num_heads = 10
embed_dim= 512
model = UNETR(img_shape=tuple(roi_size), input_dim=4, output_dim=3,
embed_dim=embed_dim, patch_size=16, num_heads=num_heads,
ext_layers=(3, 6, 9, 12), norm='instance',
base_filters=16,
dim_linear_block=2048).to(device)
Ainda não sei por que a normalização da instância funciona muito bem com os conjuntos de dados INENS e Multi-Model, mas funciona! O ponto é que temos nosso modelo de 49,7 milhões de parâmetros pronto para ser treinado.
Vamos usar o DICE Perda combinada com entropia cruzada e faça um ciclo de treinamento simples:
import torch.nn as nn
from monai.losses import DiceLoss, DiceCELoss
loss_function = DiceCELoss(to_onehot_y=False, sigmoid=True)
optimizer = torch.optim.AdamW(model.parameters(), lr=1e-4, weight_decay=1e-5)
max_epochs = 180
val_interval = 5
best_metric = -1
best_metric_epoch = -1
epoch_loss_values = ()
for epoch in range(max_epochs):
print(f"epoch {epoch + 1}/{max_epochs}")
model.train()
epoch_loss = 0
step = 0
for batch_data in train_loader:
step += 1
inputs, labels = (
batch_data("image").to(device),
batch_data("label").to(device),
)
optimizer.zero_grad()
outputs = model(inputs)
loss = loss_function(outputs, labels)
loss.backward()
optimizer.step()
epoch_loss += loss.item()
epoch_loss /= step
epoch_loss_values.append(epoch_loss)
print(f"epoch {epoch + 1} average loss: {epoch_loss:.4f}")
Comparação de linha de base: UNET
No entanto, a maior questão aqui é o quão bom esse modelo pode ser executado. Por esse motivo, precisamos de uma linha de base forte! O que é melhor do que o bem configurado UNET usado no tutorial inicial?
Também comparei minha implementação com a implementação do UNRET da Monai. Por que? Porque não haveria significado se eu corresponda ao desempenho do Sonhos Bastura e ainda tem desempenho inferior à implementação oficial. Afinal, mudei meu código para refletir as mudanças arquitetônicas do código oficial. E, de fato, vi ganhos enormes no desempenho em comparação com uma implementação simplista da figura do artigo.
from monai.networks.nets import UNet
model = UNet(
dimensions=3,
in_channels=4,
out_channels=3,
channels=(16, 32, 64, 128, 256),
strides=(2, 2, 2, 2),
num_res_units=2,
).to(device)
Vamos ver o primeiro do número:
| Modelo | épocas | Coeficiente médio de dados. |
| Sonhos (linha de base) | 170 | 76,6 % |
| UNRET (Auto-Attention-CV) | 180 | 76,9 % |
| UNRETR (monai) | 180 | 76,1 % |
Para rastrear o treinamento, medimos a perda de treinamento da perda de dados e da entropia cruzada. Também relatamos os coeficientes de dados para os 3 rótulos (canais), o núcleo tumoral (TC), o tumor inteiro (WT) e o aprimoramento do tumor (CE).
Abaixo, você pode ver essas métricas durante o treinamento:
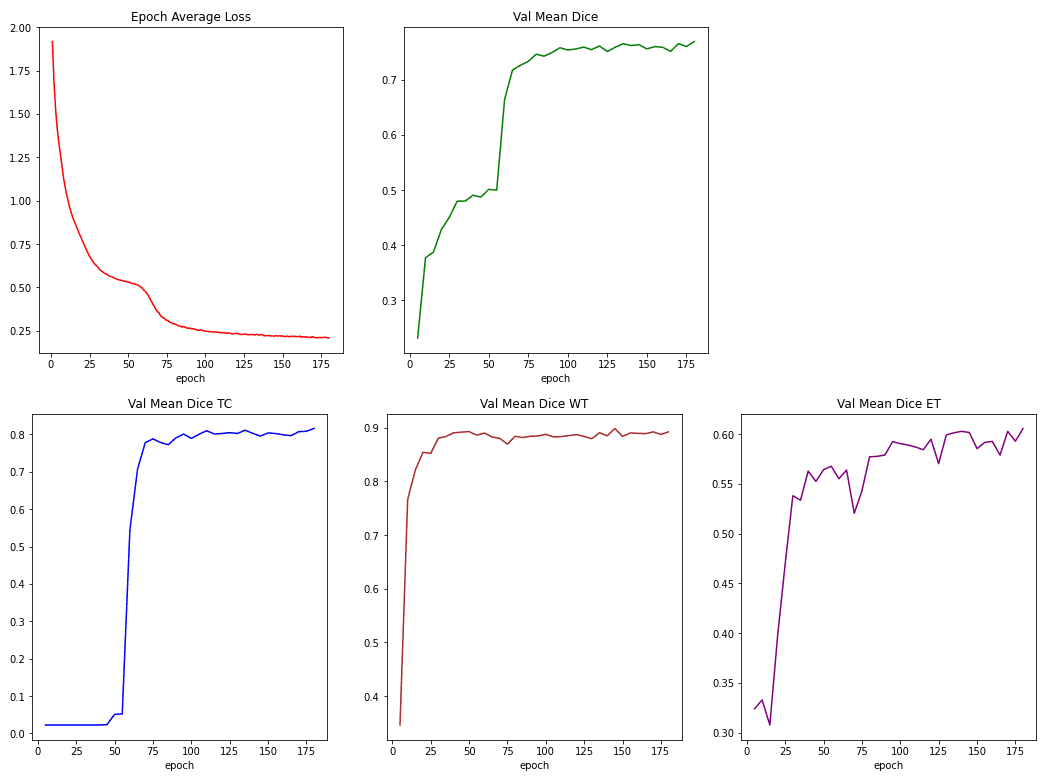
Fonte: Imagem do autor com base no caderno
Finalmente, pode -se ver os resultados comparando o mapa de segmentação de saída em comparação com a verdade do solo:
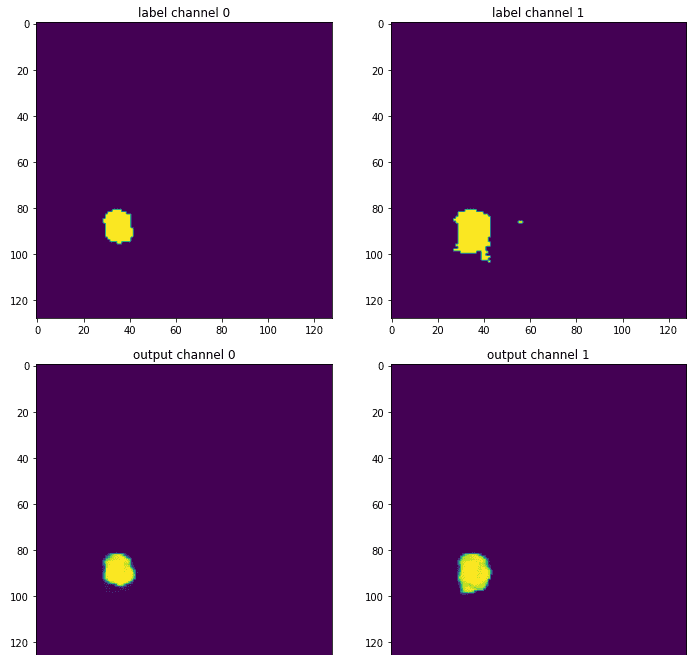
Fonte: Imagem do autor com base no caderno
O canal da área necrótica é omitido porque essa fatia em particular quase não tinha ocorrências desse rótulo. Esta ilustração é apenas uma fatia do meio do mapa de segmentação 3D, então certamente não é o quadro inteiro. Ainda assim, fornece a sensação de como o modelo treinado fornece uma versão mais sufocada da etiqueta original, que foi anotada por um radiologista especialista. Porque, como sempre, as redes neurais adoram espaços de otimização suave.
Conclusão e preocupações
Ainda não estou convencido pelo desempenho de transformadores em imagens médicas 3D. Acredito que métodos mais avançados e outras contribuições acompanham. No entanto, admito que é o primeiro trabalho interessante que desafia as arquiteturas unet bem configuradas, que são a opção preferida nessas tarefas.
A partir da análise acima, acho crucial destacar também que o aspecto mais importante para obter um bom desempenho, aqui o coeficiente de dados, é os pipelines de pré -processamento e transformação de dados. É exatamente por isso que vejo inovação limitada no imagem médica mundo em termos de modelagem de aprendizado de máquina e trabalho mais promissor sobre otimização de processamento de dados. Isso por si só não causa nenhum problema, mas me deixa muito suspeito quando um novo artigo sai e reivindica uma nova arquitetura. Como as comparações geralmente não são justas nos domínios de nicho em que trabalhei como imagens médicas.
Como sempre, obrigado pelo seu interesse na IA e fique atento a mais. Estamos orgulhosos de compartilhar com você nosso livro sobre “Aprendizado profundo em produção”, Que ensina como colocar seu modelo em produção e ampliá -lo. O apoio da comunidade (como o compartilhamento de mídia social) é sempre apreciado.
* Divulgação: Observe que alguns dos links acima podem ser links de afiliados e, sem nenhum custo adicional, ganharemos uma comissão se você decidir fazer uma compra depois de clicar.

Luis es un experto en Ciberseguridad, Computación en la Nube, Criptomonedas e Inteligencia Artificial. Con amplia experiencia en tecnología, su objetivo es compartir conocimientos prácticos para ayudar a los lectores a entender y aprovechar estas áreas digitales clave.

